几乎在所有的生物过程中,环状蛋白质低聚物都起着关键性的作用,它们占蛋白质数据库(PDB)中所有解析结构的近30%。环状蛋白质低聚物具有较为广泛的应用,从小分子结合和催化到纳米级组装的构建等。自蛋白质设计领域取得突破以来,这种环状蛋白结构的自主设计一直备受关注,然而目前的方法均需要事先对单体的结构进行规范化,除了参数化设计的螺旋束外,还涉及将先前表征的单体刚性对接到高阶对称结构中,然后才能进行界面优化,以将低能量传递给组装状态。近年来,深度学习的方法为进一步广泛探索天然蛋白质序列和结构之外的蛋白质结构提供了极大空间。最近,来自华盛顿大学生物化学系的D. Baker等人使用深度网络学习来生成范围极广的对称性蛋白质同源寡聚体,且只需指定原聚体的数量和原聚体长度即可实现。该研究设计了七种晶体结构、以及三种具有1550个残基和C33对称性的巨型纳米环(10 nm)的低温电子显微镜结构均与计算模型非常相似(中位均方根偏差:0.6埃);研究者得到的所有这些环形蛋白组装体都与现有已知的的结构有较大不同,该结果表面通过深度学习可以为生成具有丰富结构的新蛋白质组装体或纳米机器结构提供可能。
值得注意的是,今年初,David Baker课题组在Science上才发表了关于设计动态蛋白质复合物的研究。
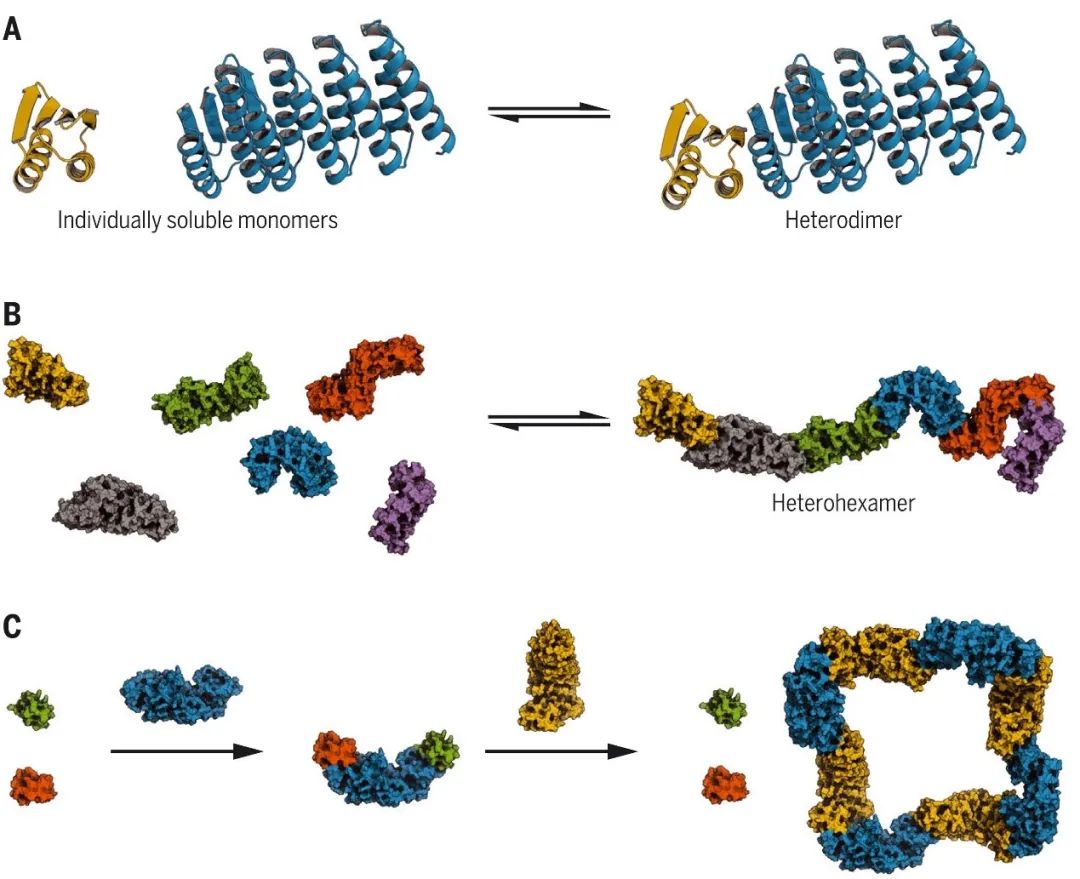
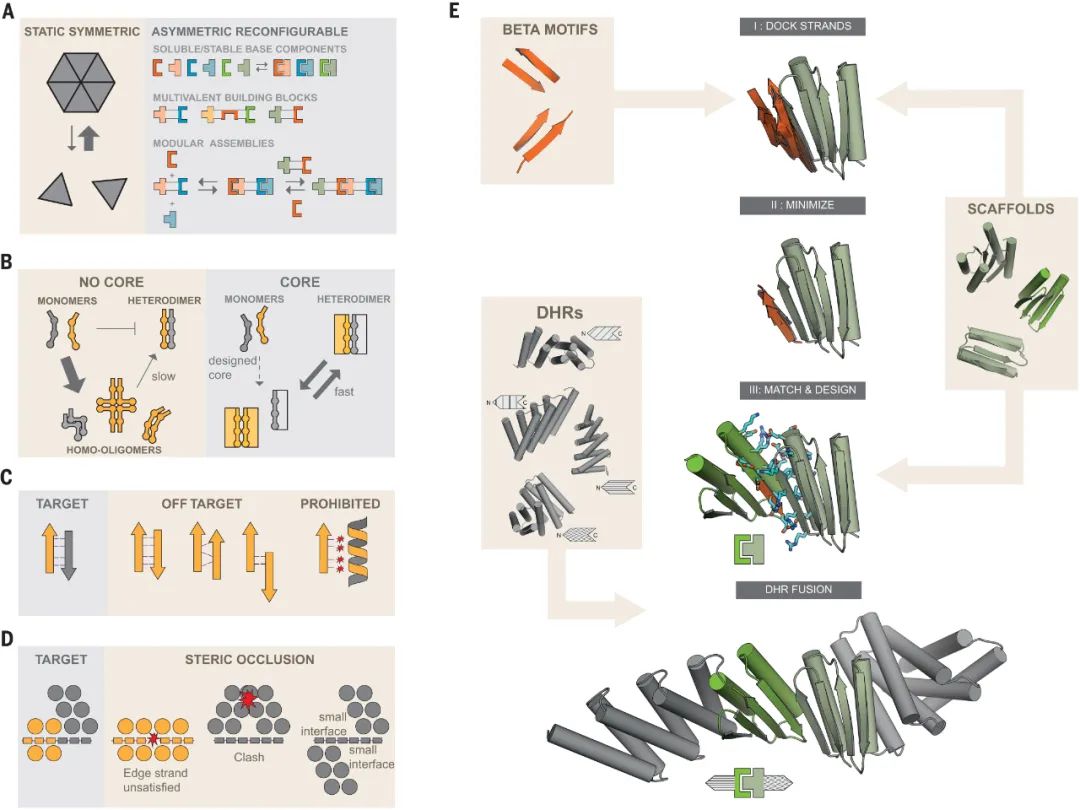
经历亚基交换的不对称多蛋白复合物在生物学中发挥着核心作用,但也给设计带来了挑战,因为这些组分不仅必须包含能够实现可逆结合的界面,而且还必须在隔离时保持稳定和良好的性能。有鉴于此,美国华盛顿大学的David Baker等研究人员,通过隐式负设计生成可重构的不对称蛋白质组件。研究人员使用隐式负设计来生成可以组装成多种复合物的 β 折叠介导的异二聚体。这些设计是稳定的、折叠的、可单独溶解的,在混合时能快速组装,并且晶体结构接近计算模型。研究人员构建了具有多达六种不同组分的线性排列的异质低聚物,支化的异质低聚物,闭合的 C4 对称双组分环,以及组装在环状同质低聚物中心枢纽上的异质低聚物,并证明这种复合物可以通过亚基交换重新配置。
本文研究的方法为设计不对称的可重构蛋白质系统提供了一般的路线。Baker D., et al. Hallucinating symmetric protein assemblies. Science. (2022).DOI: 10.1126/science.add1964https://www.science.org/doi/10.1126/science.add1964Danny D. Sahtoe, et al. Reconfigurable asymmetric protein assemblies through implicit negative design. Science, 2022.DOI:10.1126/science.abj7662https://www.science.org/doi/10.1126/science.abj7662