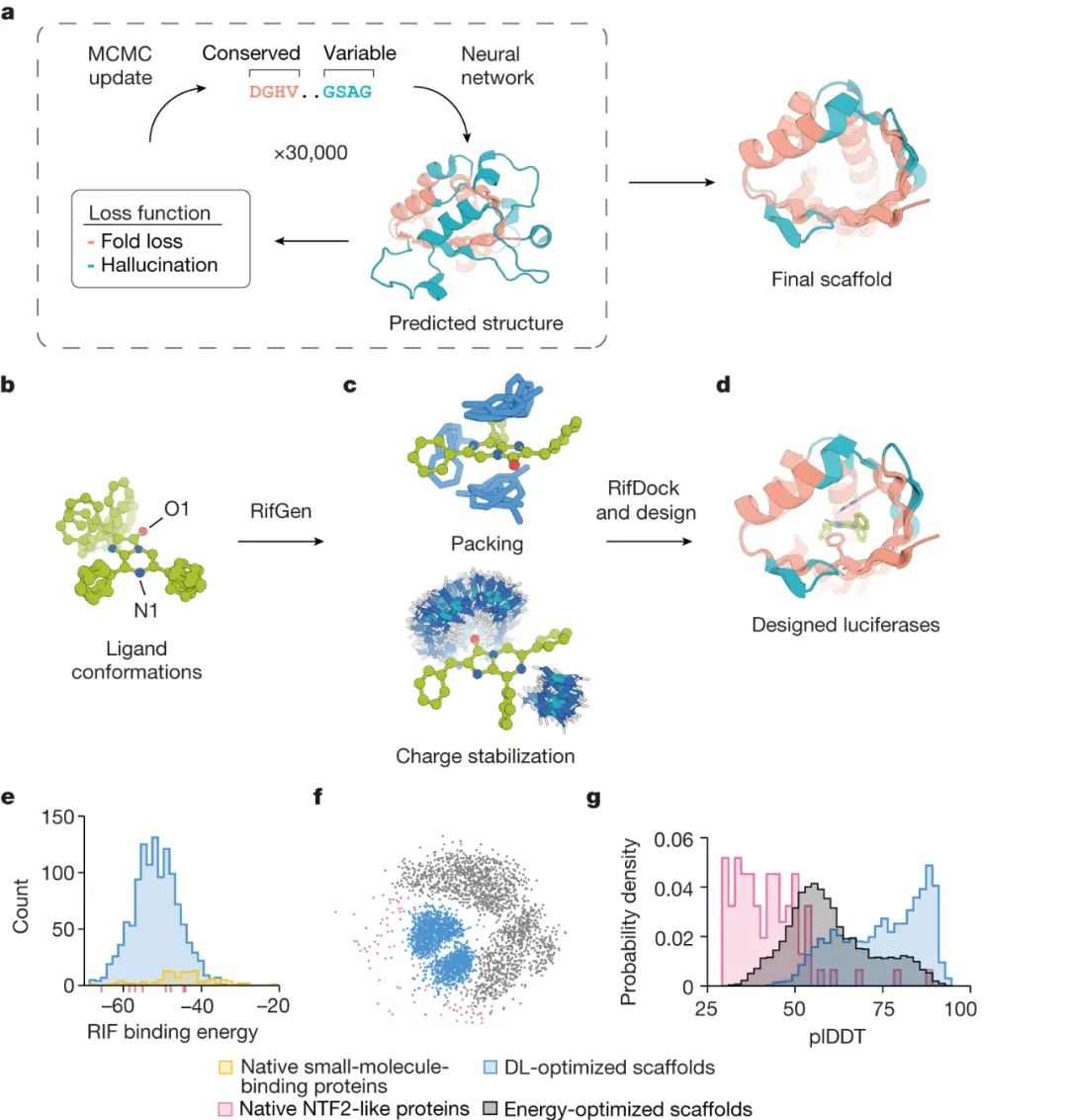
通过荧光素酶酶促氧化荧光素底物产生的生物发光被广泛用于生物医学研究中的生物测定和成像。因为不需要激发光源,所以在黑暗中产生发光光子;这导致在活体动物模型和生物样品中比荧光成像更高的灵敏度,其中自体荧光或光毒性是一个问题。然而,荧光素酶作为分子探针的开发落后于发达的荧光蛋白工具包,原因有很多:(i)很少发现天然荧光素素酶;(ii)许多已鉴定的那些需要多个二硫键来稳定结构,因此容易在哺乳动物细胞中发生错误折叠;(iii)大多数天然萤光素酶不识别具有更理想的光物理性质的合成萤光素;(iv)使用相互正交的荧光素酶-荧光素对并行进行多个过程的多重成像受到天然荧光素酶底物特异性低的限制。为了克服这些局限性,华盛顿大学David Baker、Andy Hsien-Wei Yeh等人试图使用基于“family-wide hallucination”方法从头蛋白质设计来产生小的、高度稳定的、在细胞中表达良好的、对一种底物具有特异性且不需要辅因子发挥作用的荧光素酶。

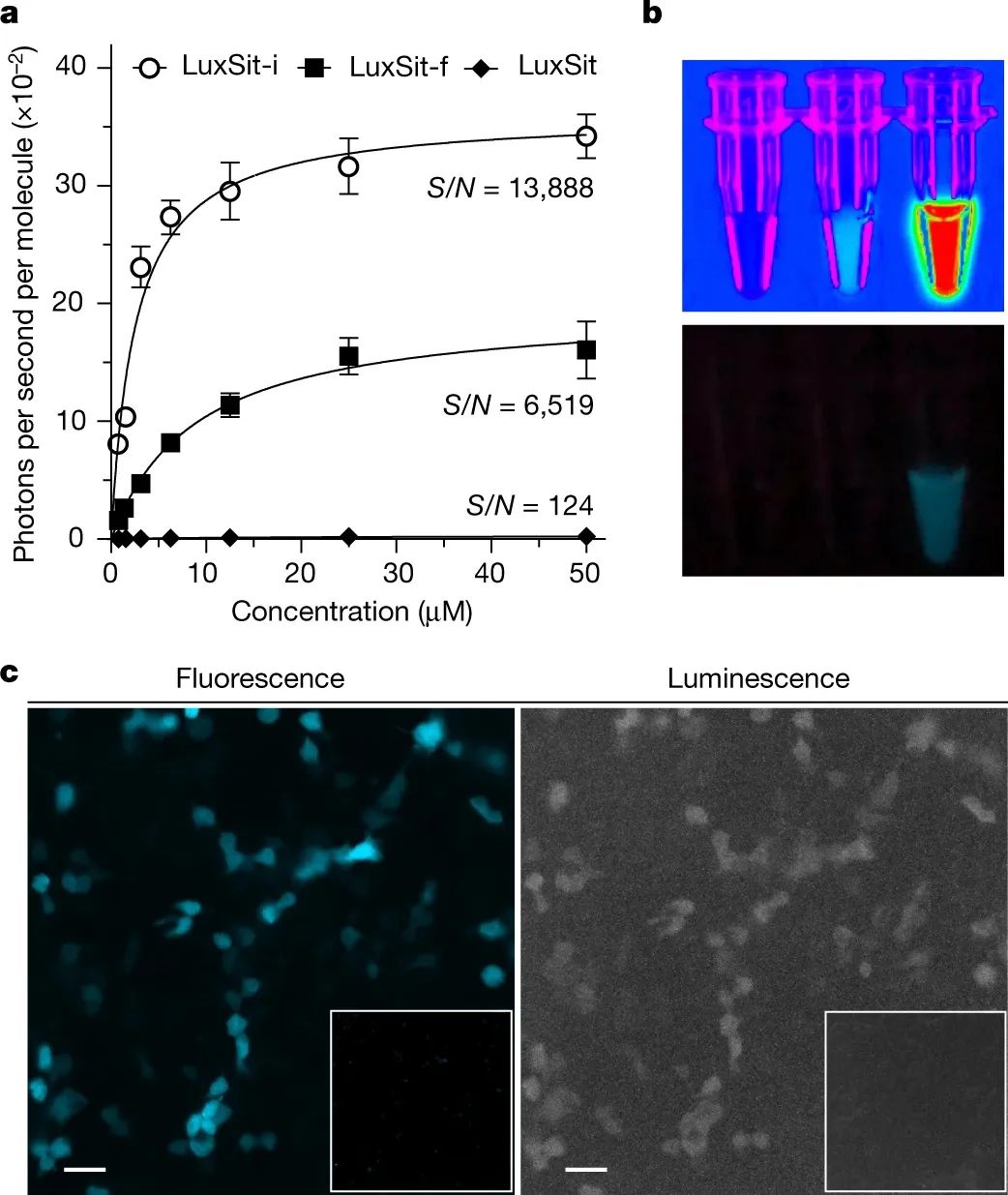
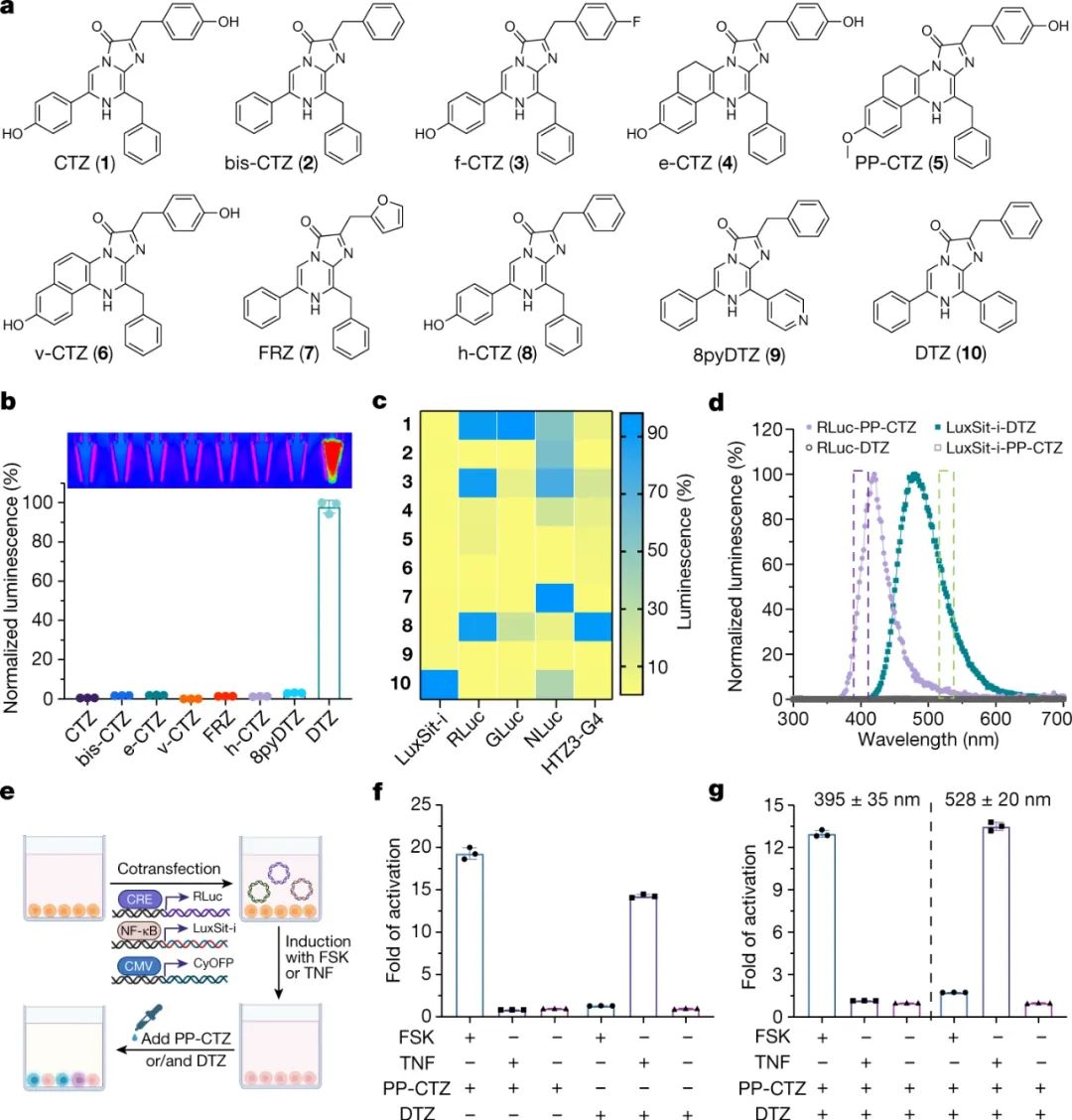
研究人员选择了一种合成的荧光素二苯噻嗪(DTZ)作为靶底物,因为它具有高量子产率、红移发射、有利的体内药代动力学以及缺乏发光所需的辅因子。为了鉴定能够容纳适合DTZ放置的口袋的蛋白质折叠,研究人员将DTZ与4000种天然小分子结合蛋白对接。研究人员发现,许多核运输因子2(NTF2)样折叠具有形状互补性和大小适合DTZ放置的结合袋,并选择了NTF2样超家族作为目标拓扑。作者产生了大量小而稳定的蛋白质支架,其具有适合DTZ的大小和形状的口袋,并具有清晰的序列结构关系,以便于随后的活性位点掺入。在从头开始的酶设计中使用深度学习使得能够产生大量具有不同口袋形状的理想化蛋白质结构和设计的编码它们的序列,克服了可用支架数量的限制。这种方法允许从头开始创建高活性和特异性的生物催化剂,这是计算酶设计的关键里程碑。所设计的酶(如LuxSit-i)的高活性和特异性使其非常适合用于生物医学以及作为酶催化机制的计算和实验研究的模型系统。本研究中使用的family-wide hallucination方法为底物结合和催化残基放置打开了几乎无限的可能性,这在反应机理和如何促进反应尚未完全理解的情况下尤为重要。除了荧光素酶催化的光发射之外,这种方法应该很容易适用于各种各样的化学反应。总之,通过使用深度学习的从头酶设计来开发高度特异性和活性的酶是计算酶设计领域的一项重大进展,并有望在生物医学和其他领域广泛应用。Yeh, A.HW., Norn, C., Kipnis, Y. et al. De novo design of luciferases using deep learning. Nature 614, 774–780 (2023). https://doi.org/10.1038/s41586-023-05696-3