蛋白质-蛋白质相互作用在几乎所有的生物过程中发挥着重要作用,包括信号转导、细胞生长和免疫防御。鉴于这些相互作用在稳态和疾病反应中的关键作用,与体内蛋白质相互作用的合成蛋白质(例如工程抗体)代表了现代医学中最具变革性的治疗方式之一。目前,大多数合成蛋白质是使用实验平台开发的,这些平台不知道这些蛋白质在何处以及如何与它们的靶标结合。尽管计算设计方法取得了进步,但预测与靶标相互作用的区域的氨基酸序列仍然是结构生物学中最具挑战性的问题之一。然而,对蛋白质-蛋白质相互作用的深入理解、结构数据可用性的增加以及机器学习的进步为改进设计蛋白质-蛋白质相互作用的方法奠定了基础。
长期以来,科学家们一直认为蛋白质通过其表面的互补区域相互作用,就像相邻的拼图块一样。然而,蛋白质-蛋白质界面从来都不是完全互补的,蛋白质表面由于原子运动而不断变化。此外,这些表面的形状和它们形成的互补化学相互作用(如氢键)的类型千差万别,这使得识别互补的表面尤其具有挑战性。
2020年,洛桑联邦理工学院Bruno E. Correia等人在Nature Methods上报道了一种称为 MaSIF 的方法,将蛋白质表面模式转化为数字阵列,可用于识别倾向于形成蛋白质-蛋白质相互作用的区域,以及与特定靶标互补的表面。
近日,Bruno E. Correia联合牛津大学Michael Bronstein等人提议可以使用这些展示来创建与生物医学相关靶标结合的新蛋白质,方法是首先识别蛋白质中的靶标位点,然后从大型蛋白质表面数据库中识别互补表面。成果发表在Nature上。

为实现这一靶标,研究人员从公共数据库中可用的蛋白质结构中构建了小蛋白质片段(或种子)的数据库。选择这些种子以形成与感兴趣的靶标位点互补的区域的基础,并随后使用计算设计将其整合到更大的蛋白质单元中。
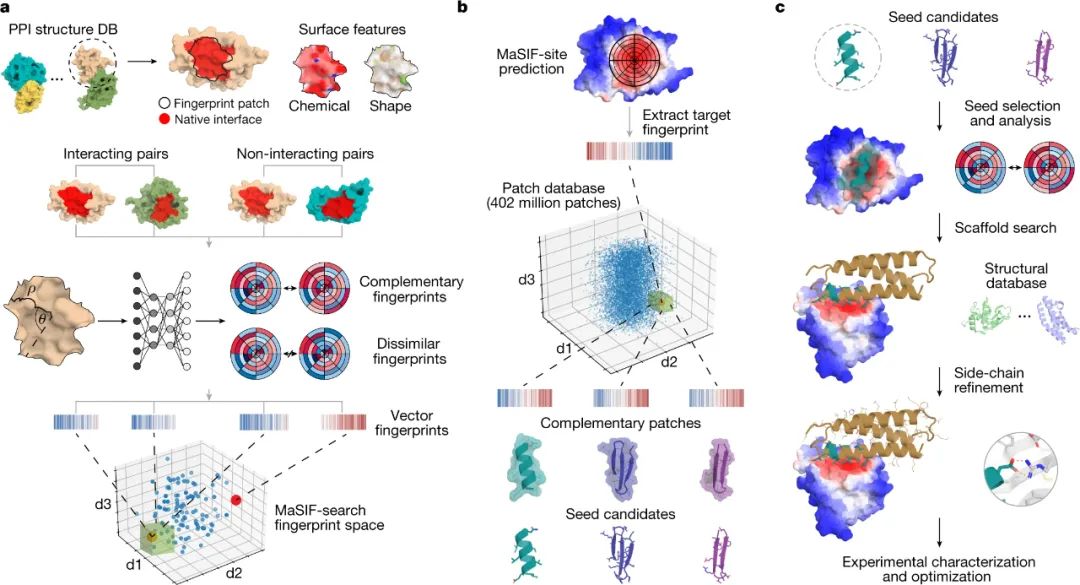
图|以表面为中心的全新位点特异性蛋白质结合剂设计
通过这种方法,研究人员为 SARS-CoV-2 病毒的刺突蛋白和癌症免疫治疗的三个重要靶点(PD-1、PD-L1和CTLA-4)创建了新的蛋白结合剂,它们的结合亲和力都接近传统抗体。此外,所有设计的蛋白质都参与了它们被开发的位点。总的来说,该管道可以很容易地推广到几个具有治疗意义的蛋白质靶点,并且可以直接从计算机生成蛋白质结合剂。
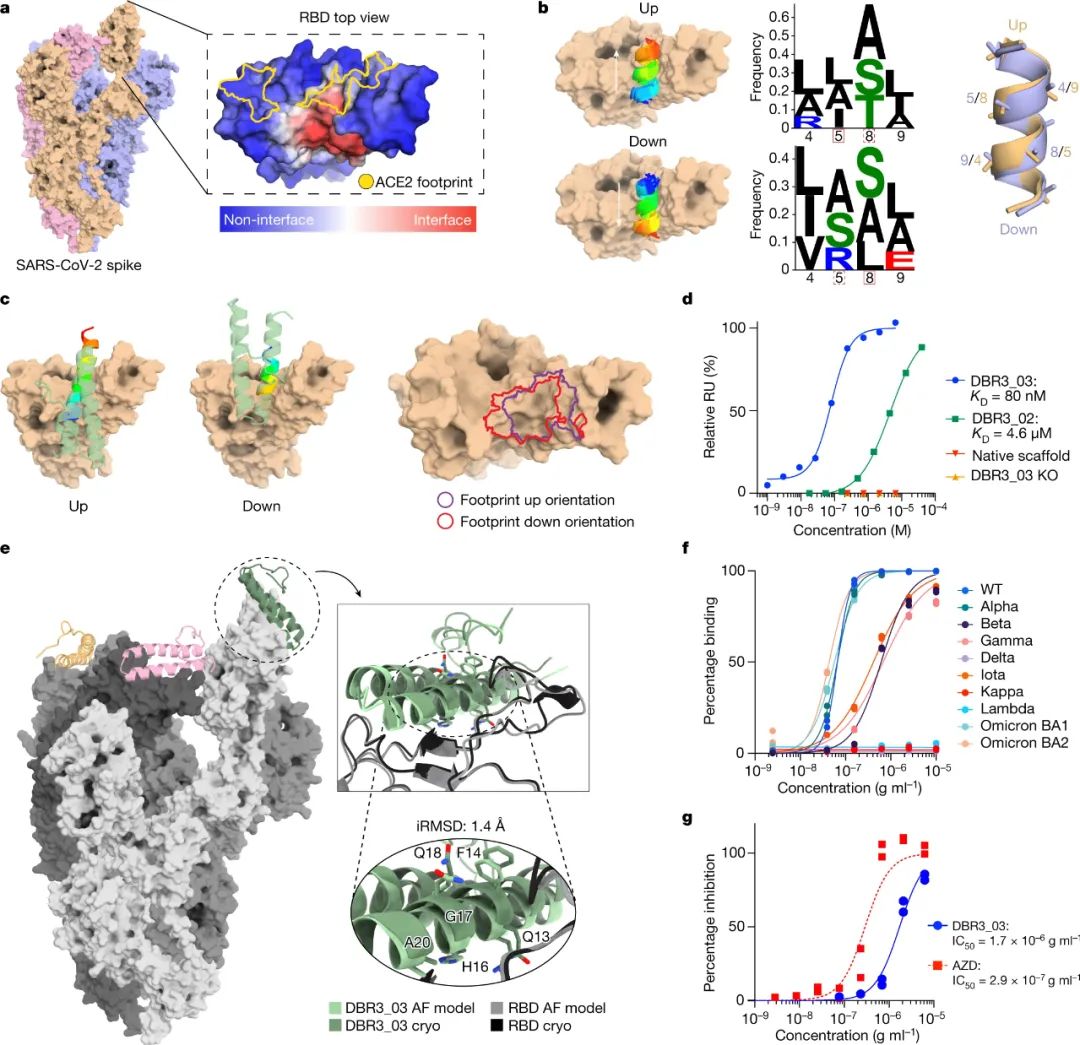
图|针对RBD的SARS-CoV-2 型结合剂的设计和优化
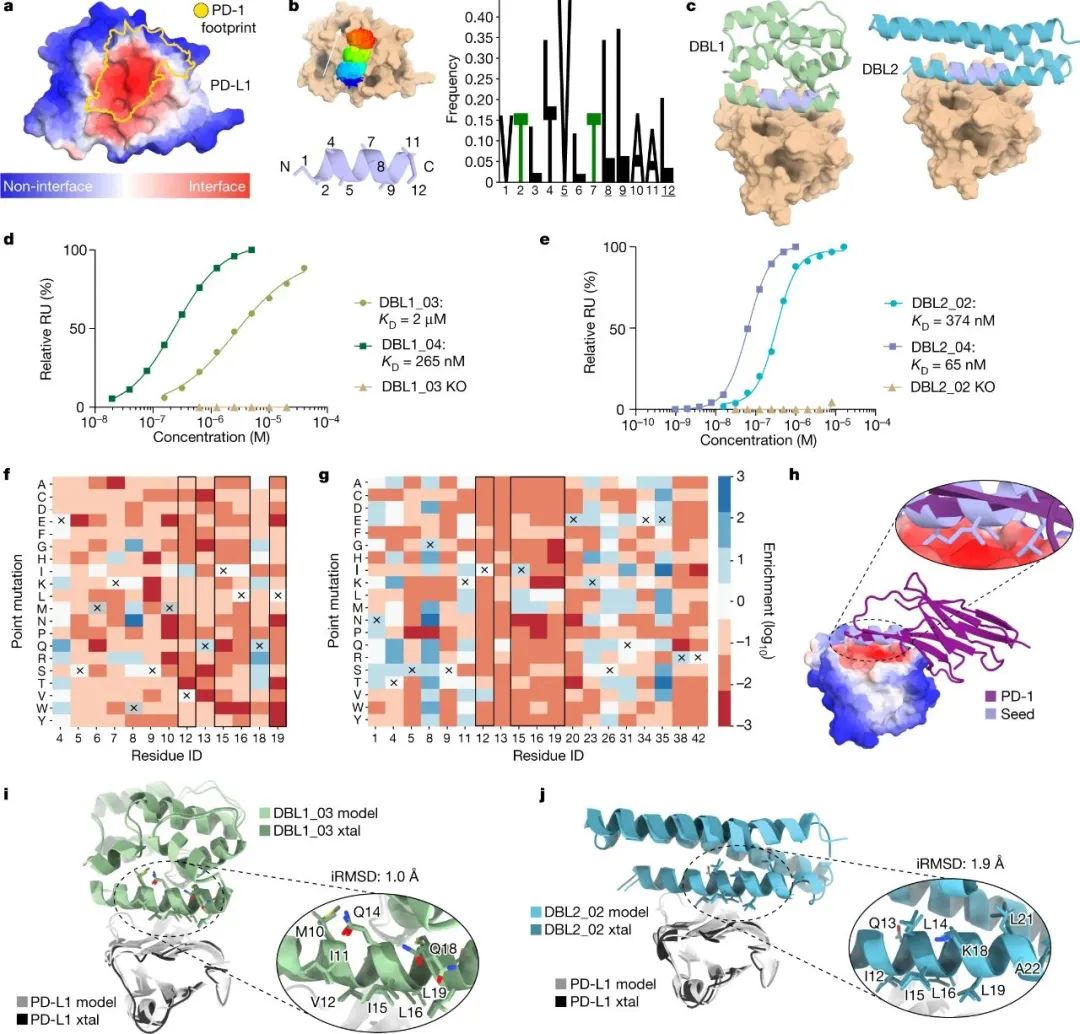
图|针对平面的PD-L1结合剂的从头设计和优化
总而言之,该研究为许多基础生物学和治疗应用打开了大门。科学家们可以使用该管道来创建比靶向特定位点甚至特定构象状态的抗体更小的蛋白质结合剂。可以选择位点来降低突变蛋白逃避合成蛋白结合的风险,就像世界上经历过的一些 SARS-CoV-2 变体一样,这些变体对各种 COVID-19 疫苗不太敏感。编码合成蛋白质的基因可以插入细胞疗法中,例如针对癌症进行修饰的免疫细胞。
当前的管道依赖于3D蛋白质种子数据库,该数据库虽然很大,但并不能普遍代表所有类型的蛋白质界面。所选片段是结构化且可预测的,这与抗体等中发现的片段不同。此外,该方法不模拟蛋白质中潜在的构象变化。总体而言,当可以使用当前工具预测合成蛋白质的构象结构并且靶标处于特征明确的构象中时,它表现良好。
参考文献:
1. Gainza, P., Wehrle, S., Van Hall-Beauvais, A. et al. De novo design of protein interactions with learned surface fingerprints. Nature (2023).
https://doi.org/10.1038/s41586-023-05993-x
2. Gainza, P. et al. Nature Methods 17, 184–192 (2020).











