
特别说明:本文由学研汇技术中心原创撰写,旨在分享相关科研知识。因学识有限,难免有所疏漏和错误,请读者批判性阅读,也恳请大方之家批评指正。
原创丨彤心未泯(学研汇 技术中心)
编辑丨风云
荧光显微镜具有分子特异性,是生命科学中用于了解复杂生物系统的主要表征方法之一。超分辨率方法可以在15至20nm范围内实现细胞分辨率,但单个生物分子之间的相互作用发生在10nm 以下的长度尺度上,分子内结构的表征需要埃级分辨率。最先进的超分辨率已经实现了在某些体外条件下空间分辨率低至5 nm和定位精度为1 nm。然而,这样的分辨率并不能直接转化为细胞实验,而埃级分辨率迄今尚未得到证实。
有鉴于此,德国马普研究所Ralf Jungmann等人介绍了一种DNA条形码方法,即通过顺序成像(RESI)提高分辨率,使用现成的荧光显微镜硬件和试剂将荧光显微镜的分辨率提高到埃级。通过以>15 nm 的中等空间分辨率对稀疏目标子集进行顺序成像,证明了完整细胞中的生物分子可以实现单蛋白分辨率。通过实验解决了埃级分辨率的DNA中单个碱基的DNA骨干距离。原理验证演示中使用该方法在未处理和药物处理的细胞中原位绘制免疫疗法靶标CD20的分子排列,这为评估靶向免疫疗法的分子机制提供了可能性。这些观察表明,通过在环境条件下对整个完整细胞进行分子内成像,RESI缩小了超分辨率显微镜和结构生物学研究之间的差距,从而提供了理解复杂生物系统的关键信息。
RESI概念
广角单分子定位显微镜 (SMLM)中目标分子的定位精度最终和根本上受到每次闪烁事件收集的光子数量的限制。SMLM无法解析的两个或多个点会产生重叠的定位分布,从而排除了将定位唯一分配给各个目标的可能性。然而,如果每个定位都可以通过颜色、条形码或任何其他分子身份分配给特定目标,则它们可以按目标明确分组。作者使用 Exchange-PAINT17针对相同的目标分子介绍了这一概念的直接实现。将此实施方式称为通过顺序成像(RESI)实现的分辨率增强,以及由此产生的定位RESI定位。通过在计算机中应用RESI,证明了超分辨率的分辨率改进。
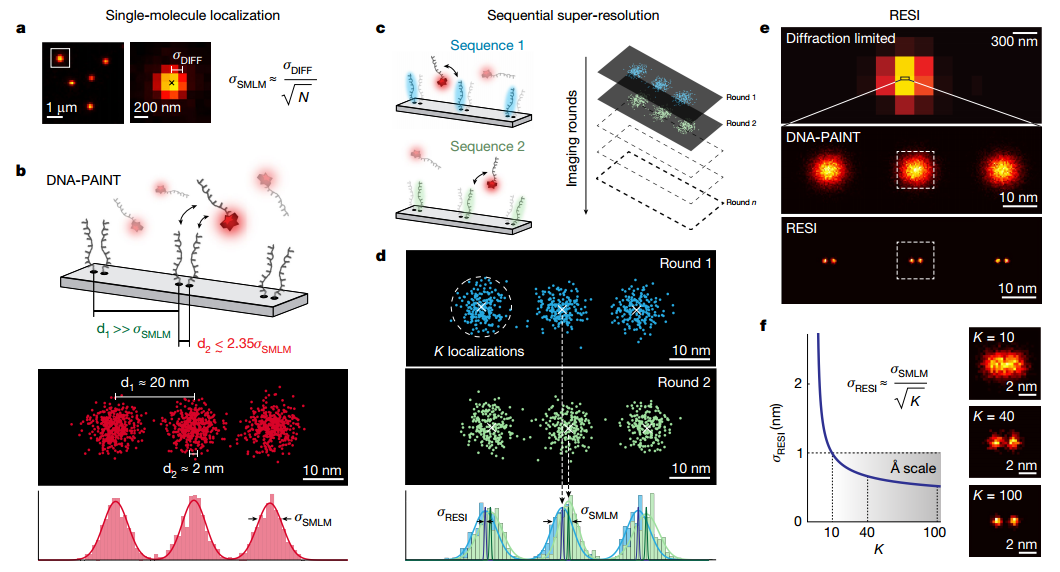
图 RESI概念
RESI 解析单核孔复合蛋白
为了证明RESI在细胞环境中的适用性,接下来对核孔复合体(NPC)的结构蛋白进行了成像。作者展示了 Nup96 分子(标记有单体增强型绿色荧光蛋白 (mEGFP))的典型衍射极限和DNA-PAINT 图像,这些分子标记有 DNA 偶联的抗GFP纳米抗体。定位组后续RESI超定位实现了定期可视化Nup96蛋白的单个复制。重建的RESI图像具有大约1nm的平均横向定位精度,比单独的 DNA-PAINT采集轮次提高了六倍。使用SMLM数据无模型方法对1217个 NPC进行了无偏3D平均,重现了Nup96 在细胞质和核环中的八重对称性以及结构平均值。借助 RESI 前所未有的空间分辨率,解决了大多数 Nup96蛋白质对的这种空间排列,这在以前是光学显微镜无法实现的。
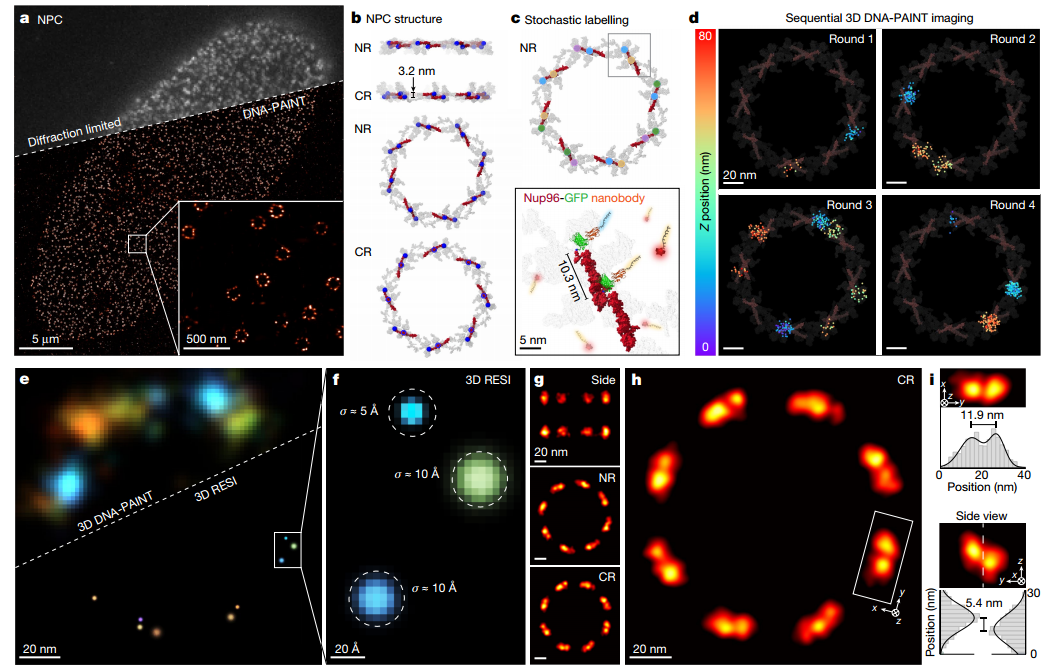
图 RESI以Ångström精度解析全细胞中的NPC蛋白
以Ångström分辨率对DNA碱基成像
为了通过RESI分析最终可实现的空间分辨率,设计了一个扁平的矩形DNA结构。RESI识别了所有DNA结构中的单链位置,在100分钟的图像采集时间内实现了这一点。作者测量了单个DNA结构中两条单链之间的距离为8.5 ± 1.7 Å,通过区分小于一纳米的结构,这证明了光学显微镜前所未有的分辨率。为了量化分辨率增益,计算了不同K值下的DNA-PAINT定位的RESI定位,证明了有效定位精度与成比例,对于平均K=254产生的平均定位精度为1.3Å。

图 RESI以Ångström分辨率解析单个DNA碱基对的距离
CD20受体组织
最后,应用RESI来研究CD20 膜受体的组织,迄今为止,原生细胞环境中的低温EM和现有的超分辨率技术都无法解决这个问题。作者应用RESI研究瞬时转染mEGFP-CD20的中国仓鼠卵巢 (CHO)细胞中CD20的分子排列,使用四轮探针交换总成像时间为4.4小时。在未经处理的细胞的衍射极限概述和 DNA-PAINT 超分辨率图像中,CD20出现均匀分布,而RTX处理的细胞表现出明显的 CD20 簇。为了定量评估未处理细胞中二聚体的存在,对DNA-PAINT和RESI数据进行了首次最近邻距离(NND)分析,证明了两种情况下的非随机分布。最后,通过与数值模拟进行比较,探讨了六聚体环状排列的存在。实验检测到的CD20簇的特征表明不存在孤立的六聚体。
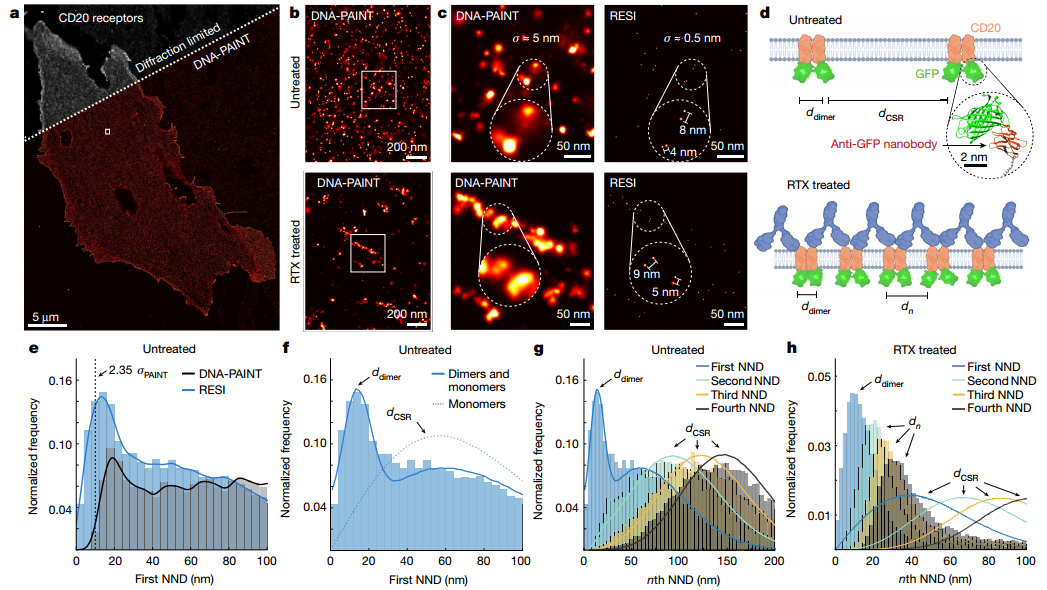
图 RESI显示药物治疗后亚纳米精度的CD20受体(重组)组织
参考文献:
Reinhardt, S.C.M., Masullo, L.A., Baudrexel, I. et al. Ångström-resolution fluorescence microscopy. Nature 617, 711–716 (2023).
DOI: 10.1038/s41586-023-05925-9
https://doi.org/10.1038/s41586-023-05925-9











