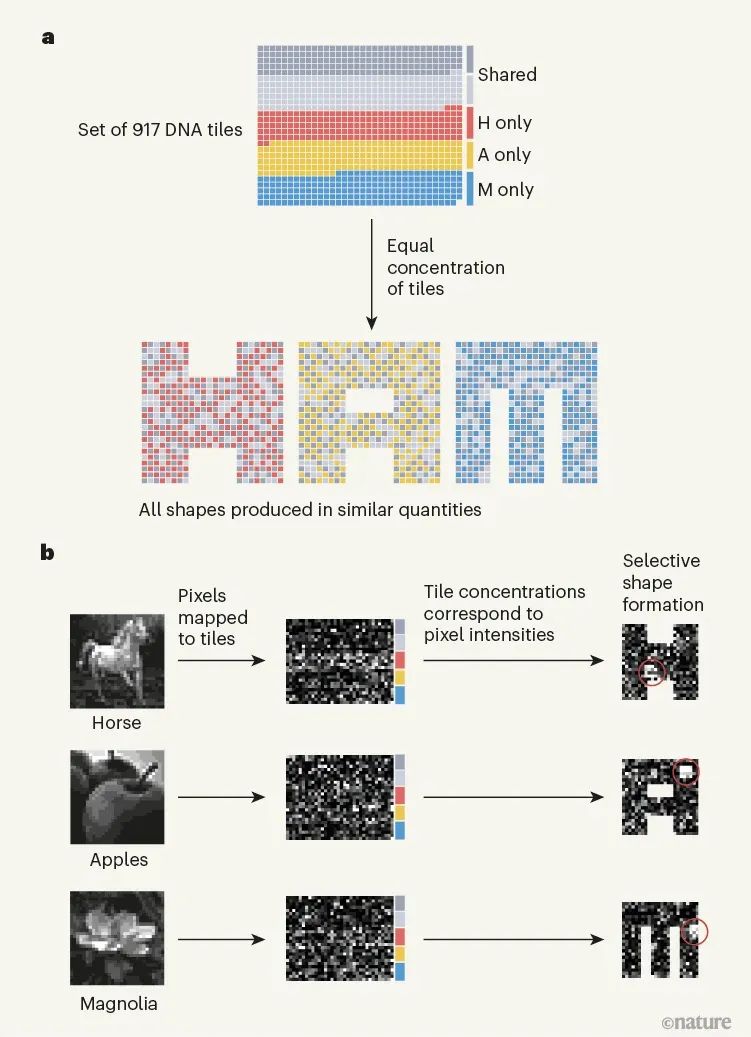
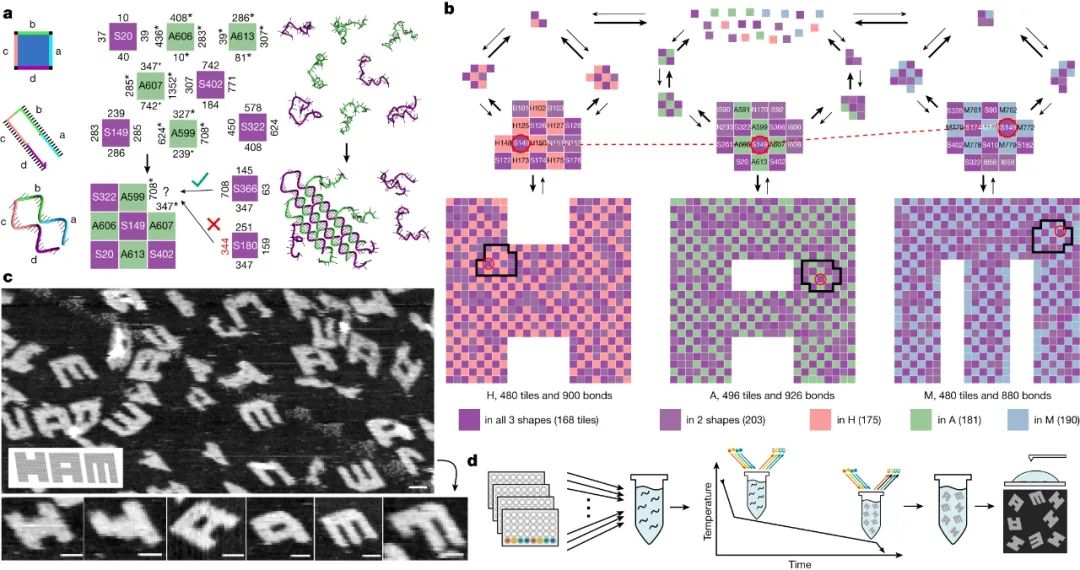
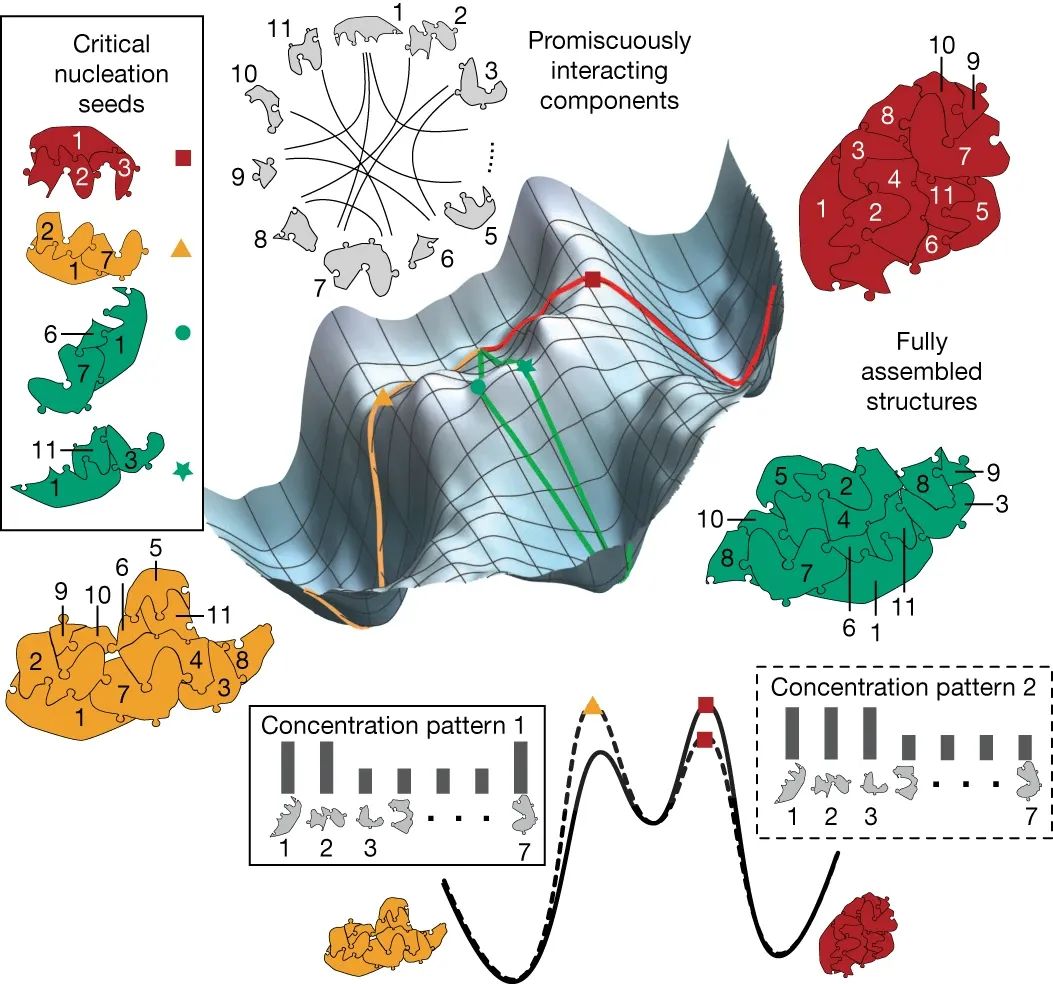
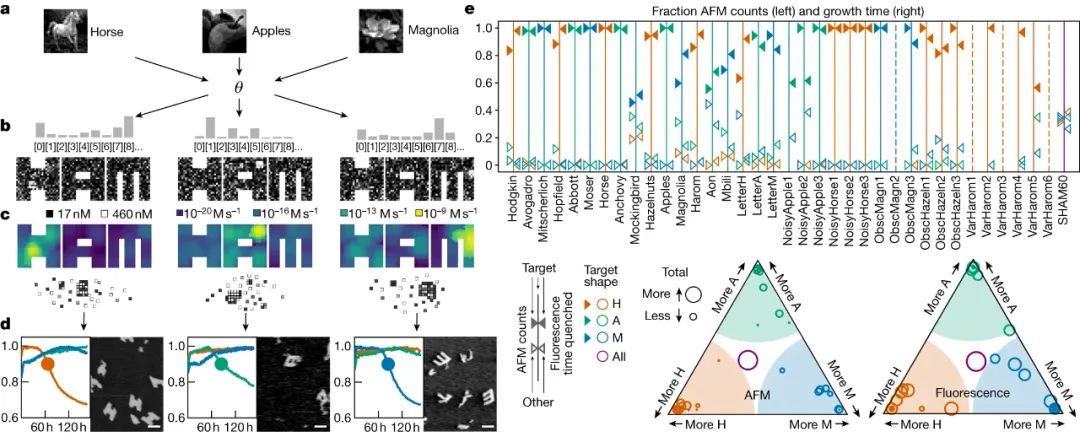
生物系统是如何处理信息的?这样的计算发生在多个尺度上,涉及分子、细胞、组织、器官等。例如,计算电路嵌入分子通路中,由大脑的神经元形成,免疫系统通过处理大量分子输入来计算免疫反应。了解这种信息处理可以解锁治疗疾病的策略,并确定生物学的基本工程原理。在人类工程系统中,计算通常在专用的处理单元中进行。相比之下,蛋白质合成、分子自组装和细胞导航等生物过程中涉及的计算往往直接嵌入生物物理过程。理解这种生物物理嵌入的计算仍然是一个关键的挑战。鉴于此,芝加哥大学Arvind Murugan、Jackson O’Brien、加州理工学院Erik Winfree、Constantine Glen Evans等人设计了一种自组装DNA系统来进行复杂的模式识别。作者的工作建立在DNA纳米技术领域的基础上,该技术使用DNA构建复杂的纳米结构,用于广泛的应用。它利用了这样一个事实,即单链DNA可以通过与具有互补序列的DNA分子结合而在溶液中自组装。一种策略是以正方形tile的形式从DNA构建块组装结构,正方形tile的设计使具有互补序列的tile边缘相互结合。这种自组装tile系统已被设计用于实现广泛的数字算法,但尚未利用这些系统探索其他复杂的计算,如模式识别。为了实现复杂的模式识别计算,研究人员设计了一组方形DNA tile,将其自组装成三种不同的形状:字母H、A和M。其中一些tile仅用于组装其中一种形状;其他的则有两三种形状。tile组的设计使得当不同的tile以相等的浓度存在于溶液中时,三种形状以相似的量形成。相比之下,每种tile的不同浓度导致了一种主要形状的形成,这取决于浓度。全tile系统是通过计算设计的,这个过程从三个形状中的每一个都开始,这些形状完全由该形状特有的tile组成,每个tile都有一组不同的边。然后,合并算法逐渐识别出可以用于多个形状的tile。该算法的每一步都从两个不同的形状中随机挑选两个tile,并试图找到一个可以同时替换它们的tile,从而相应地改变相邻的边缘。只有当生成的系统满足某些条件时,才能接受更换,以防止系统“卡”在不正确的程序集中。这些条件假设,绑定在单个边缘上的tile很容易从自组装形状脱落,而绑定在多个边缘上的tile是稳定的。当满足条件时,不正确的tile只能在单个边缘上绑定到系统;如果一个不正确的tile确实绑定了,那么随后的不正确tile也只能绑定在一条边上。经过算法的多次迭代后,获得了一个优化的系统,由 917 个tile组成,其中 371 个tile用于多个形状,具有 698 个不同的边缘。图|917种分子的多种混合物,可以从一组分子中组装成三种不同的结构下一个挑战是识别仅位于三个组装形状之一中的tile组,以形成该形状的核心。当其中一个核心在溶液中形成时,它被称为成核种子,并且其余形状的组装会以牺牲其他两个形状为代价而加速——这是“赢者通吃”的结果。这意味着可以通过选择特定的tile浓度组合(称为浓度模式)来控制从全套tile中自组装的形状,该组合可以促进该形状的成核种子的形成。研究人员使用一种算法来估计不同浓度模式形状的自组装率,从而识别出 37 个成核种子。然后使用 150 小时的退火工艺对相应的浓度模式进行实验测试,在该工艺中,tile在溶液中缓慢冷却,使其能够自组装。作者使用荧光标记来监测每个形状的组装,并使用原子力显微镜对形状进行成像。在 37 种提出的成核种子中,大约一半导致了所需形状的选择性自组装。为了展示他们的系统的信息处理能力,作者使用它根据图像中像素的阴影(灰度值)对 18 个 30×30像素的灰度图像进行分类。这个想法是通过其中一个tile的浓度来表示每个图像中每个像素的灰度值,以便生成的tile浓度模式促进指定形状(H、A 或 M)的组装。像素到tile的分配是通过计算完成的,以最大化指定形状的自组装,同时最小化竞争形状的自组装。至关重要的是,这项任务是针对所有图像同时优化的,而不是针对每个图像独立优化的。当作者通过实验测试18个图像的浓度模式时,他们观察到所需的形状确实比其他任何形状更频繁地组装,其中13个图像的选择性超过80%。换句话说,tile系统通过组装成指定的形状来识别不同的浓度模式,从而识别相应的图像。至关重要的是,该系统还处理了12个图像的降级版本。例如,当马图像的某些像素的灰度值被随机改变,从而破坏相应的浓度模式时,系统仍然可靠地形成 H 形状而不是 A 或 M,从而正确地对图像进行分类。这项工作的主要限制之一是模式识别的速度、准确性和复杂性之间的权衡。特别是,保守地选择了实验的时间尺度,以尽量减少不正确结构的形成。赢者通吃的结果表明,这些时间尺度可以大大缩短;使用由较少tile组成的较小组件也可以加快速度。因为这样的 DNA 系统可能会找到生物应用,而不是成为基于硅的计算的替代品,所以速度考虑可能不如将计算直接嵌入纳米级生物物理过程的能力重要。总的来说,最新的发现证明了如何在自组装的生物物理过程中在分子水平上编码复杂模式识别所需的计算。该研究还说明了先前关于 DNA-tile 组装的理论和实验工作如何支持复杂的新实验的设计。此外,它还展示了DNA 的可编程性以及众所周知的 DNA 碱基配对动力学和热力学如何能够设计具有 900 多个不同组件的自组装系统来执行复杂的计算。从计算的角度来看,未来工作的一个有希望的方向是进一步探索自组装系统中的模式识别与其他形式的神经计算之间的联系。研究人员在之前的工作基础上,发现了他们的tile系统和称为 Hopfield 联想记忆的神经网络模型,以及大脑中存储空间记忆的位置细胞网络之间的相似之处。进一步探索将神经计算嵌入生物物理过程的机会和局限性将是有价值的。从实验的角度来看,随着科学家设计基于蛋白质的系统和预测其生物物理相互作用的能力不断提高,该研究概述的方法可用于设计处理信息的自组装蛋白质结构。例如,具有不同功能的不同蛋白质复合物可以根据其构建模块的浓度进行自组装。更一般地说,这项工作还为未来设计嵌入生物物理过程的紧凑、稳健和可扩展的计算提供了一个概念和实验框架。Evans, C.G., O’Brien, J., Winfree, E. et al. Pattern recognition in the nucleation kinetics of non-equilibrium self-assembly. Nature 625, 500–507 (2024).https://doi.org/10.1038/s41586-023-06890-z