北京时间10月9日下午5点45分许,2024年诺贝尔化学奖揭晓。瑞典皇家科学院决定将2024年诺贝尔化学奖的一半授予大卫·贝克(David Baker),以表彰他在“计算蛋白质设计”方面的贡献,并将另一半授予戴密斯·哈萨比斯(Demis Hassabis)和约翰·M·詹伯(John M. Jumper),以表彰他们在“蛋白质结构预测”方面的贡献。下面,我们主要介绍一下我们熟悉的来自华盛顿大学的David Baker近期的研究,供读者阅读~
1. Nature:设计的内吞诱导蛋白降解靶标并放大信号

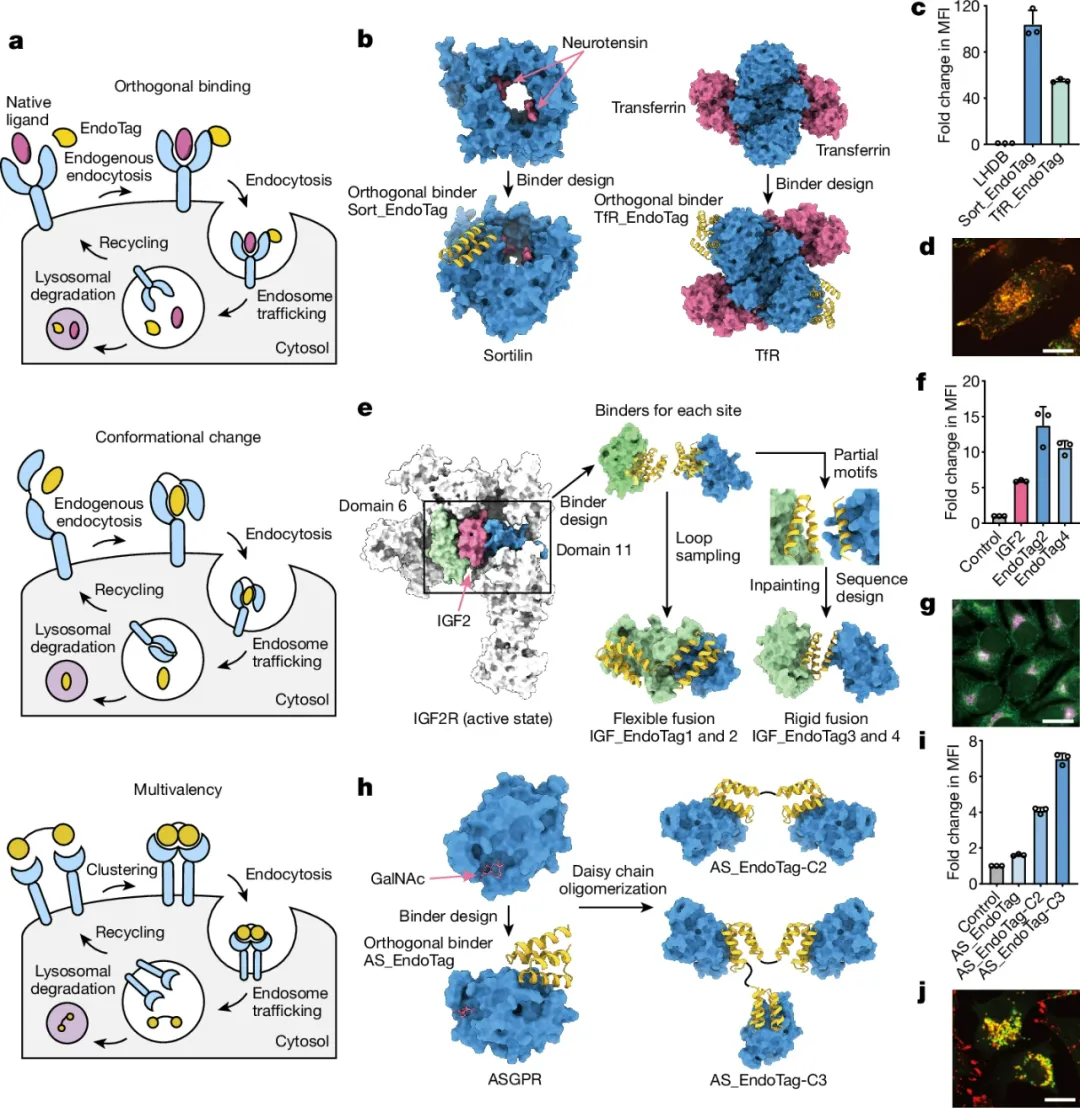
内吞作用和细胞表面受体的溶酶体运输可以通过内源性配体触发。治疗方法如溶酶体靶向嵌合体1,2(LYTACs)和细胞因子受体靶向嵌合体3(KineTACs)利用这一点,通过将修改后的天然配体与目标结合蛋白融合,来靶向特定蛋白质进行降解。尽管这些方法很强大,但它们可能受到与天然配体竞争的限制,以及化学修饰的要求,这些要求限制了遗传编码能力,并可能使制造过程复杂化,而且,更一般地说,可能没有通过给定受体刺激内吞作用的天然配体。
于此,华盛顿大学David Baker等人描述了针对内吞触发结合蛋白(EndoTags)的计算设计方法,这些方法克服了这些挑战。
研究人员为胰岛素样生长因子2受体(IGF2R)和无唾液酸糖蛋白受体(ASGPR)、分选蛋白和转铁蛋白受体提出了EndoTags,并展示了将这些标签与可溶性或跨膜目标蛋白结合物融合,可以导致溶酶体运输和目标降解。由于这些受体在不同组织中的分布不同,不同的EndoTags可以实现对不同组织的降解靶向。与PD-L1抗体融合的EndoTag在小鼠肿瘤模型中的疗效显著高于单独的抗体。
EndoTags的模块化和遗传编码能力实现了更高特异性靶向降解的AND门控制,以及从工程细胞中局部分泌降解剂。通过促进内吞作用,EndoTag融合通过近100倍的信号增强,通过工程配体-受体系统。EndoTags作为靶向降解诱导剂、内吞依赖途径的信号激活剂以及靶向抗体-药物和抗体-RNA偶联物的细胞摄取诱导剂具有相当大的治疗潜力。
参考文献:
Huang, B., Abedi, M., Ahn, G. et al. Designed endocytosis-inducing proteins degrade targets and amplify signals. Nature (2024).
https://doi.org/10.1038/s41586-024-07948-2
2. Nature:使用深度学习从头设计萤光素酶

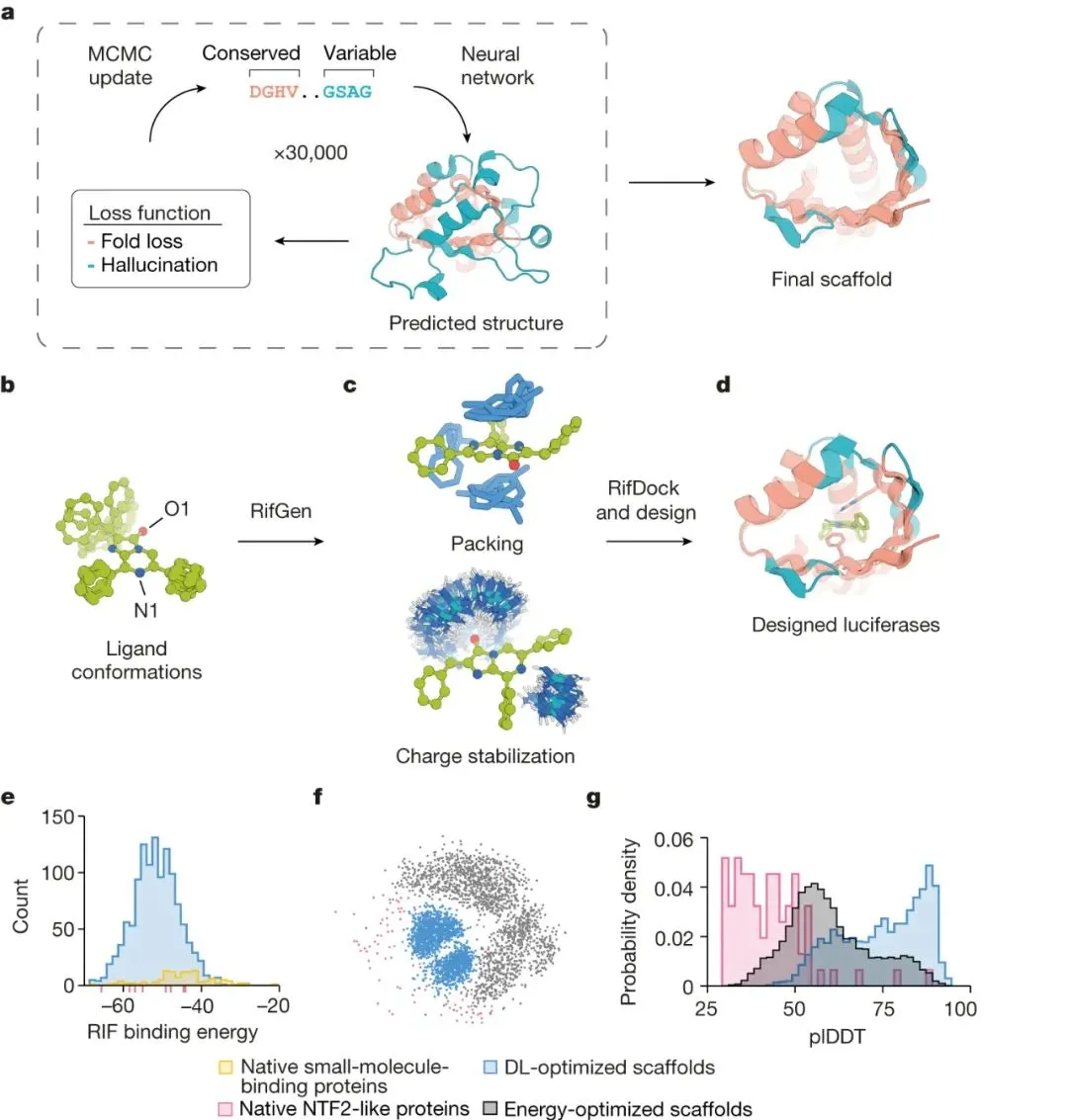
荧光素酶作为分子探针的开发落后于发达的荧光蛋白工具包,原因有很多:(i)很少发现天然荧光素素酶;(ii)许多已鉴定的那些需要多个二硫键来稳定结构,因此容易在哺乳动物细胞中发生错误折叠;(iii)大多数天然萤光素酶不识别具有更理想的光物理性质的合成萤光素;(iv)使用相互正交的荧光素酶-荧光素对并行进行多个过程的多重成像受到天然荧光素酶底物特异性低的限制。
为了克服这些局限性,华盛顿大学David Baker、Andy Hsien-Wei Yeh等人试图使用基于“family-wide hallucination”方法从头蛋白质设计来产生小的、高度稳定的、在细胞中表达良好的、对一种底物具有特异性且不需要辅因子发挥作用的荧光素酶。研究人员选择了一种合成的荧光素二苯噻嗪(DTZ)作为靶底物,因为它具有高量子产率、红移发射、有利的体内药代动力学以及缺乏发光所需的辅因子。
Yeh, A.HW., Norn, C., Kipnis, Y. et al. De novo design of luciferases using deep learning. Nature 614, 774–780 (2023).
https://doi.org/10.1038/s41586-023-05696-3
3. Nature Nanotechnology:pH响应自组装螺旋蛋白丝的从头设计

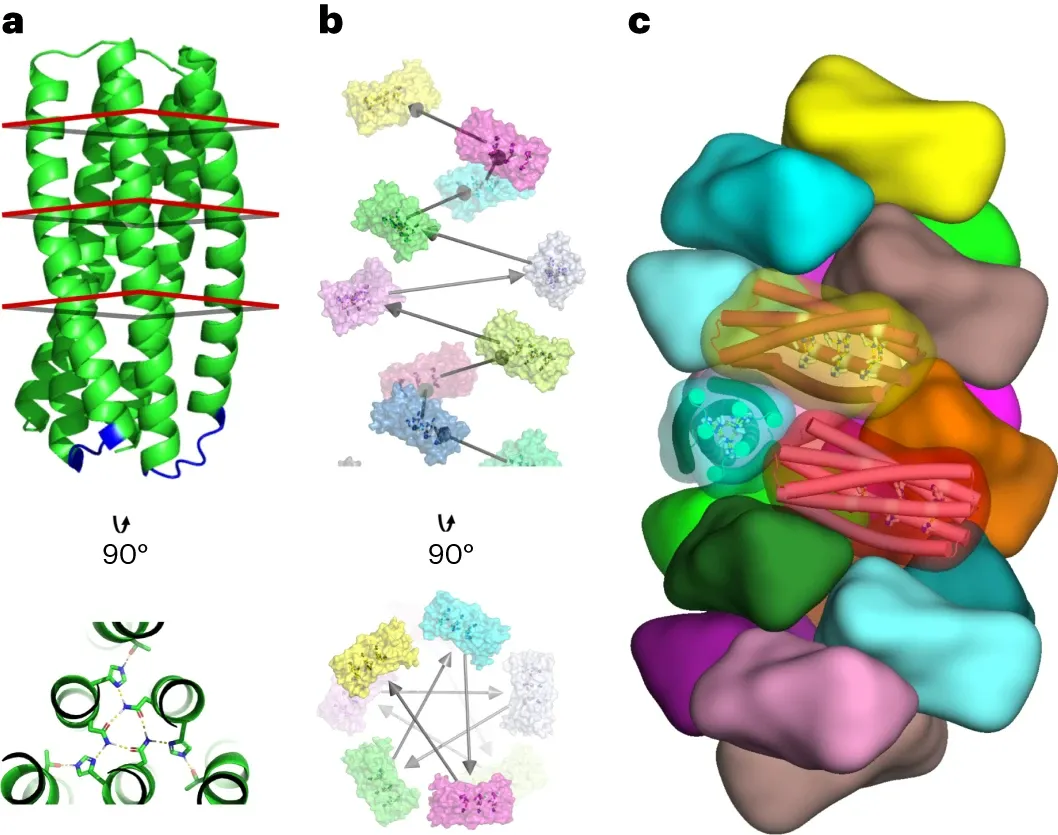
华盛顿大学David Baker、Hao Shen等研究人员描述了一种全新的pH响应性蛋白纤维的设计。这些纤维由含有六个或九个埋藏组氨酸残基的亚基组成,在中性pH下组装成微米尺度的有序纤维。通过冷冻电子显微镜(cryo-EM)确定了优化设计的结构,其与计算设计模型几乎一致,无论是亚基内部几何形状还是亚基在纤维中的堆积方式。
研究人员通过计算设计方法成功创造了能够在特定pH变化下自组装和解组装的蛋白纤维。他们设计了含有多个埋藏组氨酸残基的亚基,这些残基在pH变化时改变其质子化状态,从而触发纤维的组装和解组装。通过筛选和表达18个不同的设计,研究人员发现DpHF7和DpHF18能够在降低pH值时解组装,并在pH值回升时重新组装,展示了对pH变化的敏感和可调节的响应性。
Shen, H., Lynch, E.M., Akkineni, S. et al. De novo design of pH-responsive self-assembling helical protein filaments. Nat. Nanotechnol. (2024).
https://doi.org/10.1038/s41565-024-01641-1
4. Nature Biotechnology:新型生物传感器!
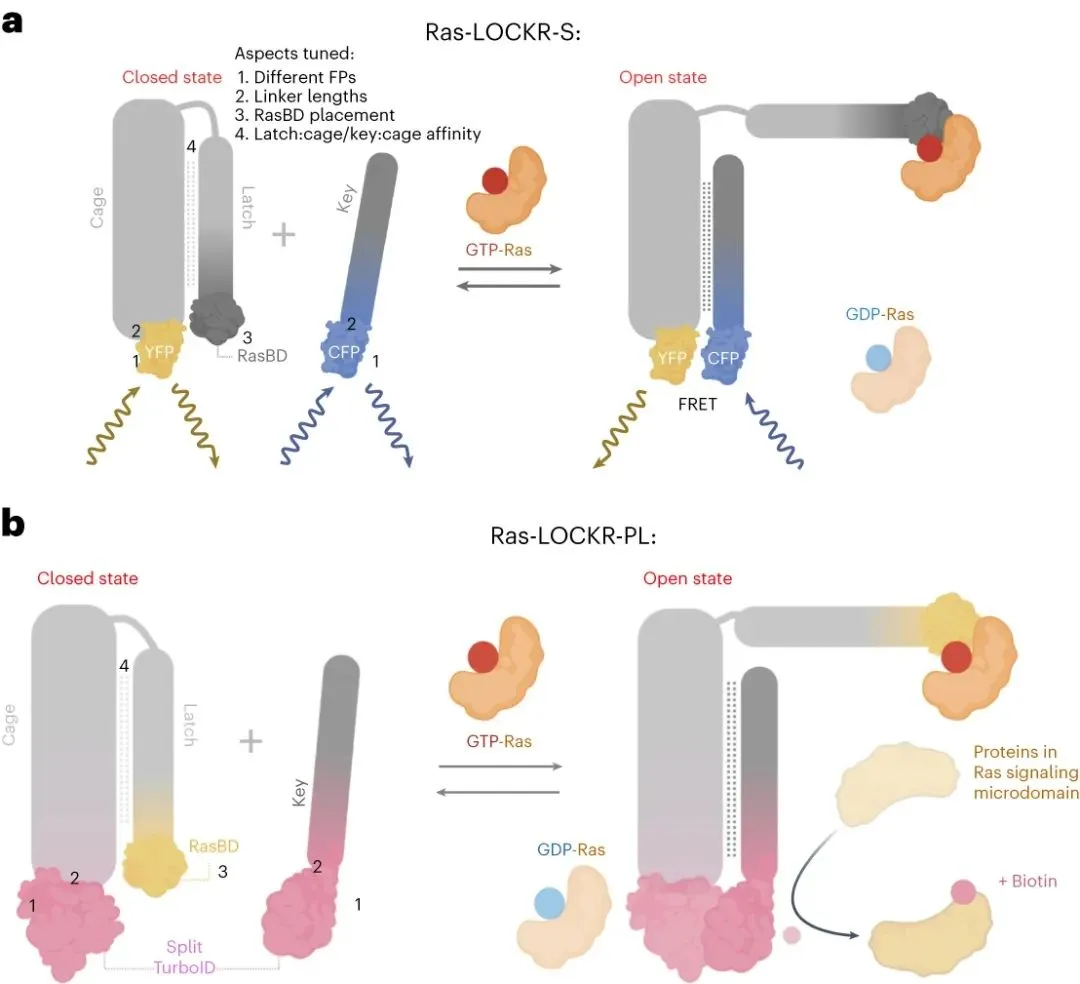
为了开发内源性活性Ras的传感器,华盛顿大学David Baker、Dustin J. Maly、Jason Z. Zhang等研究人员使用了新的传感器支架和设计方法。
对于传感器支架,研究人员使用了从头蛋白质开关LOCKR(闩锁正交笼键蛋白质)。LOCKR与天然蛋白质没有序列或结构相似性。此外,它的切换行为是清晰的,并且由于它的正交性,它可以在不影响传感器功能的情况下发生变化。
对于设计方法,研究人员使用基于Rosetta的蛋白质设计算法和AlphaFold结构预测来调整LOCKR的切换(从闭合的、自动抑制的状态转变为Ras-GTP依赖的开放状态,其中读出结构域接近),以匹配内源性活性Ras的生理相关浓度范围。利用这些策略,研究人员开发了两种传感器:一种是基于荧光的传感器,该传感器基于距离依赖性Förster共振能量转移的变化来测量Ras的活性(命名为Ras-LOCKR-S),另一种是通过生物素化活性Ras周围的附近蛋白质来表征Ras环境的基于分裂邻近标记器的传感器(命名为Ras-LOCKR-PL)。这些传感器足够灵敏以检测内源性活性Ras,与活细胞兼容以使Ras活性能够随时间测量,并且可以定位于不同的细胞区室以使Ras-GTP能够以亚细胞分辨率检测。
Zhang, J.Z., Nguyen, W.H., Greenwood, N. et al. Computationally designed sensors detect endogenous Ras activity and signaling effectors at subcellular resolution. Nat Biotechnol (2024).
https://doi.org/10.1038/s41587-023-02107-w
5. Nature Chemistry:由可延伸蛋白质复合物制成的精确图案化纳米纤维

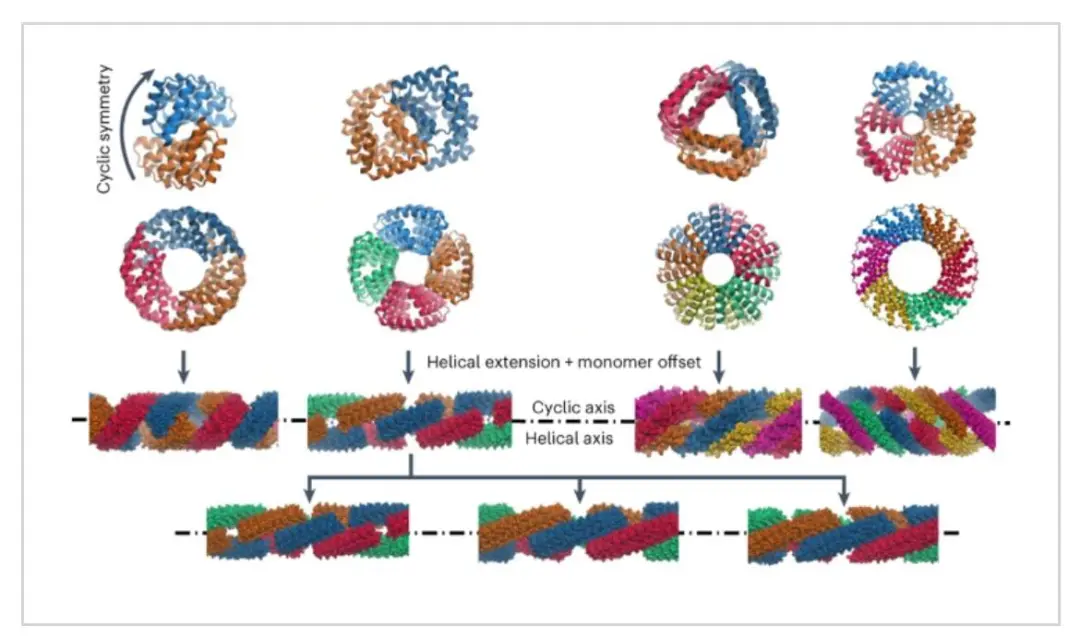
具有环状和超螺旋对称轴的分子系统在材料设计方面具有巨大优势,因为它们可以通过改变组成单体的长度来延长或缩短。在蛋白质中,α螺旋盘绕线圈具有对称、可扩展的结构,但其受到螺旋原聚体相对固定的几何形状和柔性的限制。在这里,华盛顿大学David Baker报道了由可延伸蛋白质复合物制成的精确图案化纳米纤维。
作者描述了一种产生具有重合C2至C8和超螺旋对称轴的模块化和刚性重复蛋白质低聚物的方法,该低聚物可以通过重复传播进行扩展。
在这些构建块中,作者发现可以通过引入亲水性表面片段来系统设计各种无界纤维,从而迫使单体发生交错。这种纤维的几何形状可以通过改变单体中重复单元的数量和亲水性片段的位置来精确调节。
参考文献:
Neville P. Bethel et.al Precisely patterned nanofibres made from extendable protein multiplexes Nature Chemistry 2023
DOI: 10.1038/s41557-023-01314-x
https://doi.org/10.1038/s41557-023-01314-x
6. Nature Materials:三维蛋白质晶体的精确计算设计

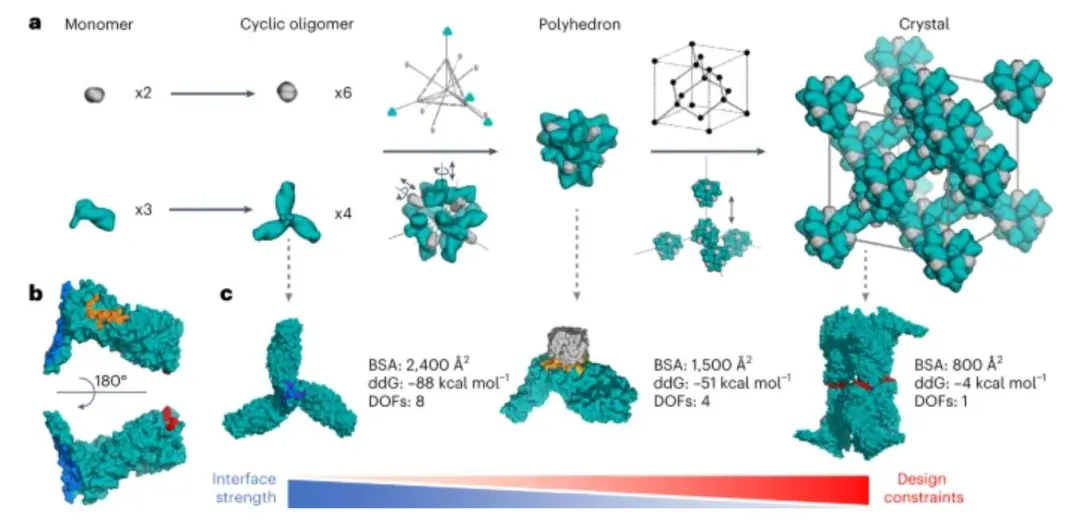
通过蛋白质-蛋白质界面上精确设计的侧链-侧链相互作用来编程蛋白质结晶极具挑战性。在这里,华盛顿大学David Baker开发了一种通用的计算方法,用于以原子精度设计具有预先指定的晶格结构的三维蛋白质晶体。
作者设计了三对低聚物,它们可以单独纯化,混合后自发地自组装成>100 µm三维晶体。这些晶体的结构与计算设计模型几乎相同,在整体结构和特定的蛋白质-蛋白质相互作用方面都非常对应。
晶体晶胞的尺寸可以被系统地重新设计,同时保持空间群对称性和整体结构,晶体具有极高的多孔性和高度稳定性。该方法能够高精度地进行蛋白质晶体的计算设计,所设计的蛋白质晶体在其初级序列中编码了结构和组装信息,为生物材料工程提供了强大的平台。
参考文献:
Zhe Li et.al Accurate computational design of three-dimensional protein crystals Nature Materials 2023
DOI: 10.1038/s41563-023-01683-1
https://doi.org/10.1038/s41563-023-01683-1
7. Science:可灵活设计的对称性蛋白质组装

几乎在所有的生物过程中,环状蛋白质低聚物都起着关键性的作用,它们占蛋白质数据库(PDB)中所有解析结构的近30%。环状蛋白质低聚物具有较为广泛的应用,从小分子结合和催化到纳米级组装的构建等。自蛋白质设计领域取得突破以来,这种环状蛋白结构的自主设计一直备受关注,然而目前的方法均需要事先对单体的结构进行规范化,除了参数化设计的螺旋束外,还涉及将先前表征的单体刚性对接到高阶对称结构中,然后才能进行界面优化,以将低能量传递给组装状态。近年来,深度学习的方法为进一步广泛探索天然蛋白质序列和结构之外的蛋白质结构提供了极大空间。
最近,来自华盛顿大学生物化学系的D. Baker等人使用深度网络学习来生成范围极广的对称性蛋白质同源寡聚体,且只需指定原聚体的数量和原聚体长度即可实现。
该研究设计了七种晶体结构、以及三种具有1550个残基和C33对称性的巨型纳米环(10 nm)的低温电子显微镜结构均与计算模型非常相似(中位均方根偏差:0.6埃);
研究者得到的所有这些环形蛋白组装体都与现有已知的的结构有较大不同,该结果表面通过深度学习可以为生成具有丰富结构的新蛋白质组装体或纳米机器结构提供可能。

Baker D., et al. Hallucinating symmetric protein assemblies. Science. (2022).
DOI: 10.1126/science.add1964
https://www.science.org/doi/10.1126/science.add1964
8. Science:通过隐式负设计生成可重构的不对称蛋白质组件
经历亚基交换的不对称多蛋白复合物在生物学中发挥着核心作用,但也给设计带来了挑战,因为这些组分不仅必须包含能够实现可逆结合的界面,而且还必须在隔离时保持稳定和良好的性能。有鉴于此,美国华盛顿大学的David Baker等研究人员,通过隐式负设计生成可重构的不对称蛋白质组件。
研究人员使用隐式负设计来生成可以组装成多种复合物的 β 折叠介导的异二聚体。
这些设计是稳定的、折叠的、可单独溶解的,在混合时能快速组装,并且晶体结构接近计算模型。
研究人员构建了具有多达六种不同组分的线性排列的异质低聚物,支化的异质低聚物,闭合的 C4 对称双组分环,以及组装在环状同质低聚物中心枢纽上的异质低聚物,并证明这种复合物可以通过亚基交换重新配置。
本文研究的方法为设计不对称的可重构蛋白质系统提供了一般的路线。

参考文献:
Danny D. Sahtoe, et al. Reconfigurable asymmetric protein assemblies through implicit negative design. Science, 2022.
DOI:10.1126/science.abj7662
https://www.science.org/doi/10.1126/science.abj7662
9. Nature:有利于简化蛋白质结合蛋白的设计过程的新方法
除了目标三维结构之外,不使用其他信息设计与目标蛋白表面特定位点结合的蛋白质仍具有挑战性。有鉴于此,美国华盛顿大学的David Baker等研究人员,仅使用目标结构设计蛋白质结合蛋白。
研究人员提出了一种解决蛋白设计的方案,首先对蛋白质表面选定区域可能结合模式的大空间进行广泛探索,然后在最有希望的结合模式附近加强搜索。研究人员通过从头设计将蛋白质与12种不同形状和表面特性的蛋白质靶标结合,证明了其具有广泛的适用性。
生物物理特性表明,所有小于65个氨基酸的结合剂是超稳定的,并且经过实验优化后,它们可与靶标蛋白以纳摩尔到皮摩尔的亲和力结合。研究人员成功解析了五种结合剂-靶标配合物的晶体结构,这五种结构都非常接近相应的计算设计模型。
近50万个计算设计和数十万个点突变体的实验数据提供了有关该方法优势、局限性以及对目前蛋白质-蛋白质相互作用理解的详细反馈,并可用于对这两种方法进行改进。
本文研究的方法可对治疗和诊断应用中各种蛋白质上的感兴趣位点进行针对性设计。

参考文献:
Longxing Cao, et al. Design of protein binding proteins from target structure alone. Nature, 2022.
DOI:10.1038/s41586-022-04654-9
https://www.nature.com/articles/s41586-022-04654-9











