 通过蛋白质-蛋白质界面上精确设计的侧链-侧链相互作用来编程蛋白质结晶极具挑战性。在这里,华盛顿大学David Baker开发了一种通用的计算方法,用于以原子精度设计具有预先指定的晶格结构的三维蛋白质晶体。
通过蛋白质-蛋白质界面上精确设计的侧链-侧链相互作用来编程蛋白质结晶极具挑战性。在这里,华盛顿大学David Baker开发了一种通用的计算方法,用于以原子精度设计具有预先指定的晶格结构的三维蛋白质晶体。
本文要点:
1) 作者设计了三对低聚物,它们可以单独纯化,混合后自发地自组装成>100 µm三维晶体。这些晶体的结构与计算设计模型几乎相同,在整体结构和特定的蛋白质-蛋白质相互作用方面都非常对应。
2) 晶体晶胞的尺寸可以被系统地重新设计,同时保持空间群对称性和整体结构,晶体具有极高的多孔性和高度稳定性。该方法能够高精度地进行蛋白质晶体的计算设计,所设计的蛋白质晶体在其初级序列中编码了结构和组装信息,为生物材料工程提供了强大的平台。
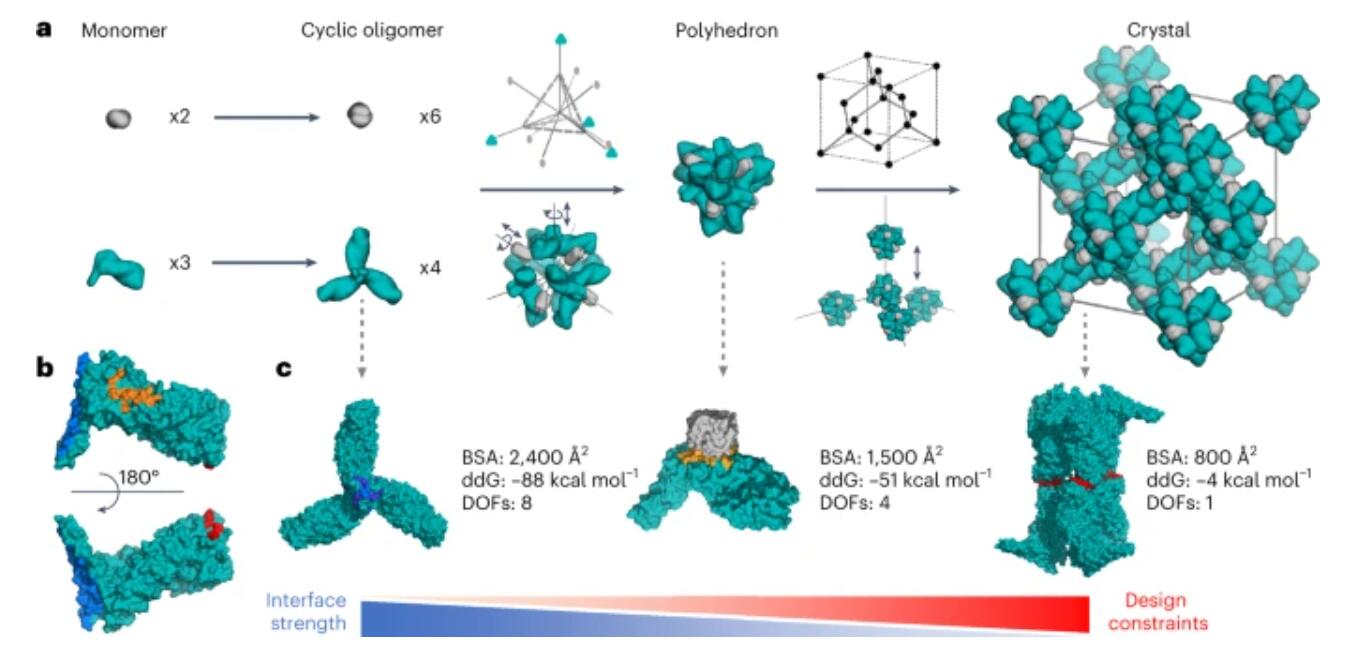
Zhe Li et.al Accurate computational design of three-dimensional protein crystals Nature Materials 2023
DOI: 10.1038/s41563-023-01683-1
https://doi.org/10.1038/s41563-023-01683-1


















