
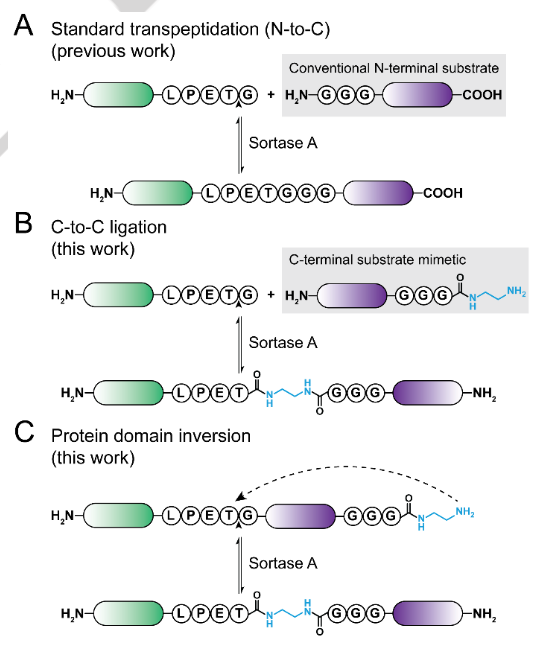
蛋白质的拓扑转换和排列作为产生新的蛋白质功能或稳定性的策略引起了人们的极大兴趣。这些努力主要受到自然发生的翻译后修饰的启发,如头尾环化、环状排列或套索状缠结。在环状排列的情况下,可以通过遗传编码来促进这种方法,或者在环化的情况下,可以通过酶处理来促进这种方法。值得注意的是,这些先前描述的策略没有改变多肽主链的方向。
昆士兰大学Fabian B. H. Rehm等描述了一种非天然的蛋白质排列,即蛋白质结构域倒位。
本文要点:
(1)
由此蛋白质的C-末端部分相对于蛋白质的N-末端部分从标准的N-to-C酶促倒位为C-to-C构型。概念上最相似的生物学过程可能是重组酶催化的DNA片段的倒置。作者使用一种工程化的分类酶A实现了这些逆转,这是一种广泛使用的转肽酶。
(2)
作者的反应在4-25°C的温和条件下有效进行,并且与完全异源产生的蛋白质底物兼容。
参考文献:
Yan Zhou, Thomas Durek, David J. Craik, Fabian B. H. Rehm, Sortase-Catalyzed Protein Domain Inversion,Angew. Chem. Int. Ed. 2024, e202316777.
DOI: 10.1002/anie.202316777
https://doi.org/10.1002/anie.202316777


















