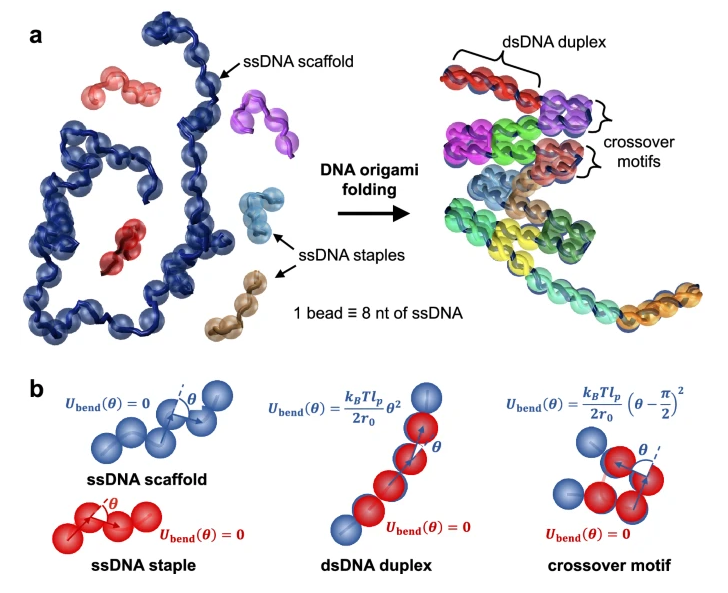
许多实验和计算工作都试图理解DNA折纸折叠,但这一过程的时间和尺度规模存在重大挑战。鉴于此,来自杜克大学的Gaurav Arya研究出了一个介观模型,该模型使用可切换的力场来捕捉单链和双链DNA基序的行为以及它们之间的转变,能够模拟大小高达几千碱基的DNA折纸的折叠。
文章要点:
1) 该研究证实,小结构的布朗动力学模拟揭示了一个分级折叠过程,包括压缩成部分折叠的前体,到结晶成最终结构,同时,该研究阐明了各种设计选择对折叠顺序和动力学的影响;
2) 此外,该研究表明,较大的结构表现出异质短纤维结合动力学和频繁的亚稳态捕获,而更容易接近的结构则表现出一级动力学和几乎无缺陷的折叠,并且,该模型为更好地理解和设计DNA纳米结构以提高产率和折叠性能提供了依据。
参考资料:
DeLuca, M., Duke, D., Ye, T. et al. Mechanism of DNA origami folding elucidated by mesoscopic simulations. Nat. Commun. (2024).
DOI:10.1038/s41467-024-46998-y
https://doi.org/10.1038/s41467-024-46998-y


















