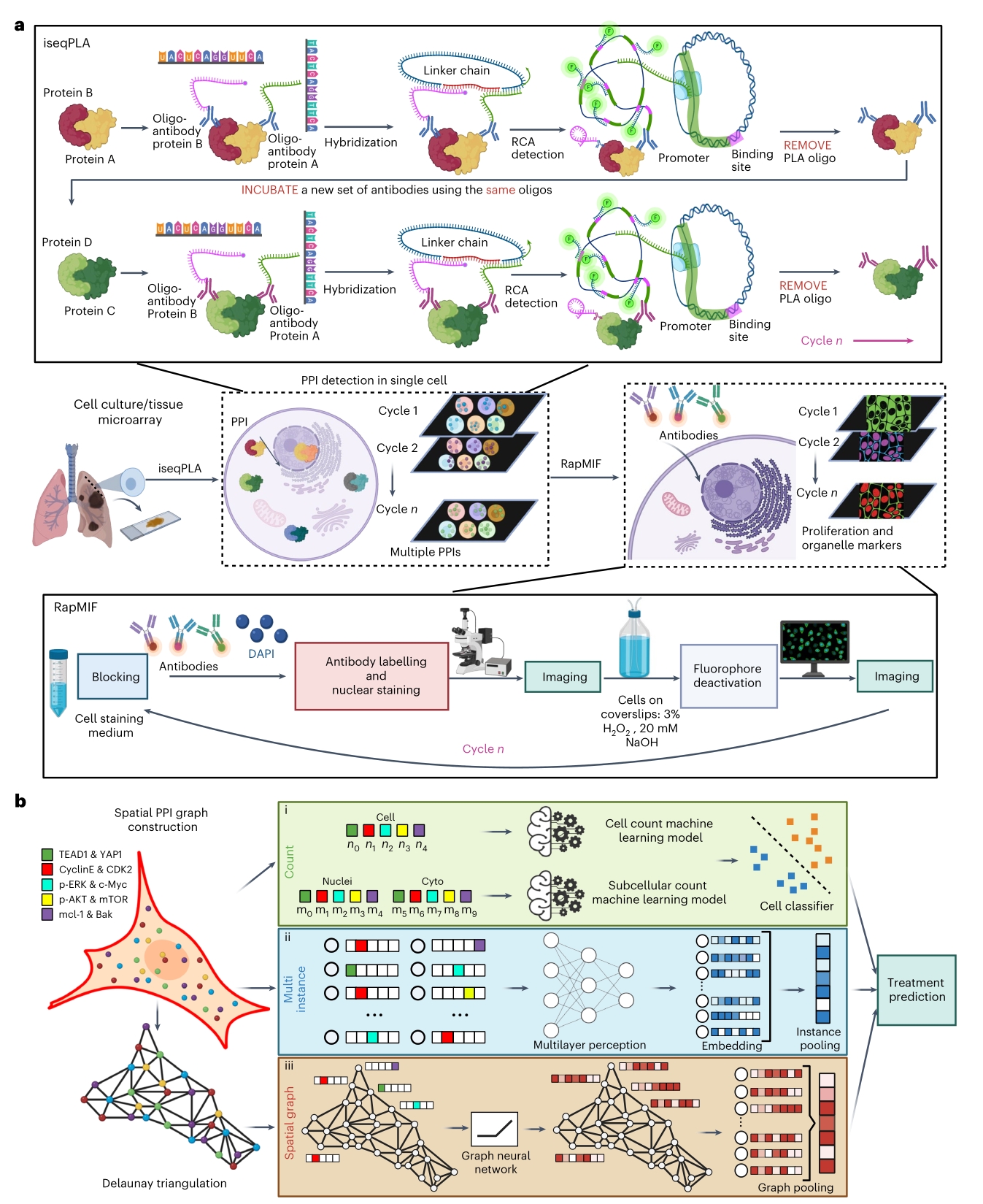
蛋白质相互作用(PPIs)调节信号通路和细胞表型,因此PPIs空间分辨动力学的可视化将揭示信号网络的激活和串扰。于此,佐治亚理工学院和埃默里大学Ahmet F. Coskun等人报告了一种方法,该方法利用顺序邻近连接分析法对PPI进行多重分析,其中多达47种蛋白质参与多信号串扰通路。
研究人员该方法应用于具有突变表皮生长因子受体的非小细胞肺癌的细胞培养和组织,然后进行常规免疫荧光,以确定PPI在亚细胞体积中的共定位,并重建PPI在酪氨酸激酶抑制剂osimertinib干扰下的亚细胞分布变化。研究还表明,编码空间分辨PPI的图卷积网络可以准确预测单个细胞的细胞处理状态。基于图的深度学习辅助的多重邻近连接分析可以深入了解PPI的亚细胞组织,从而设计靶向蛋白质相互作用组的药物。
参考文献:
Cai, S., Hu, T., Venkataraman, A. et al. Spatially resolved subcellular protein–protein interactomics in drug-perturbed lung-cancer cultures and tissues. Nat. Biomed. Eng (2024).
https://doi.org/10.1038/s41551-024-01271-x


















